INHOUDSOPGAWE:
- Outeur John Day day@howwhatproduce.com.
- Public 2024-01-30 07:26.
- Laas verander 2025-01-23 12:53.
Deur: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Hierdie MATLAB -kode is 'n program om die presiese grootte, vorm en ligging van 'n gewas in die brein -MRI -skanderings van 'n pasiënt op te spoor. Hierdie program is ontwerp om oorspronklik te werk met gewasopsporing in brein MRI -skanderings, maar dit kan ook gebruik word vir die diagnose van kanker in ander orgaanskanderings.
Die volgende instruksies beskryf eers die metodes vir beeldanalise deur die MRI -skandering te filter en op te ruim, deur binarisering, mediaanfiltering en skuifvensters. Vervolgens sal hy instruksies gee oor hoe om die tumor te isoleer met 'n vooraf gegenereerde elliptiese masker, en dit verder te filter om die omtrek van die vorm van die tumor te beskryf.
Sodra die gewas opgespoor is, sal die instruksies verder beskryf hoe u hierdie program in 'n grafiese gebruikerskoppelvlak (GUI) kan opneem. Gedurende hierdie instruksies word die toepaslike kode en lêers aangeheg om te help verduidelik hoe hierdie MRI -skandering -analise werk.
'N Paar dinge om te weet, af te laai en gereed te hê voordat u met hierdie instruksies voortgaan: 1. Maak seker dat u die nuutste weergawe van MATLAB afgelaai het. U kan R2018b hier installeer:
2. Om hierdie program uit te voer, moet u toegang hê tot MRI breinskandering lêers. Alhoewel sommige altyd uit Google -beelde gevind kan word, kan deeglike en akkurate analise uitgevoer word uit die regte beelde van verskillende lae breinskanderings vir elke pasiënt. U het toegang tot die lêers vir 20 verskillende pasiënte met glioblastoom voor en na behandeling vanaf hierdie databasis:
3. Die fokus van hierdie program en die verskillende metodes wat hierdie projek rig, word in hierdie navorsingsartikel uiteengesit:
Stap 1: Begin grafiese gebruikerskoppelvlak (GUI)
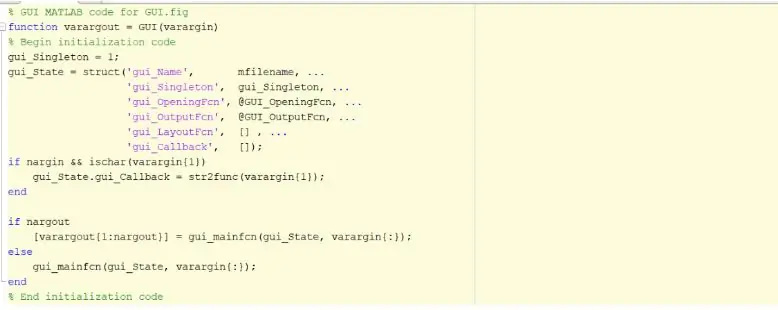
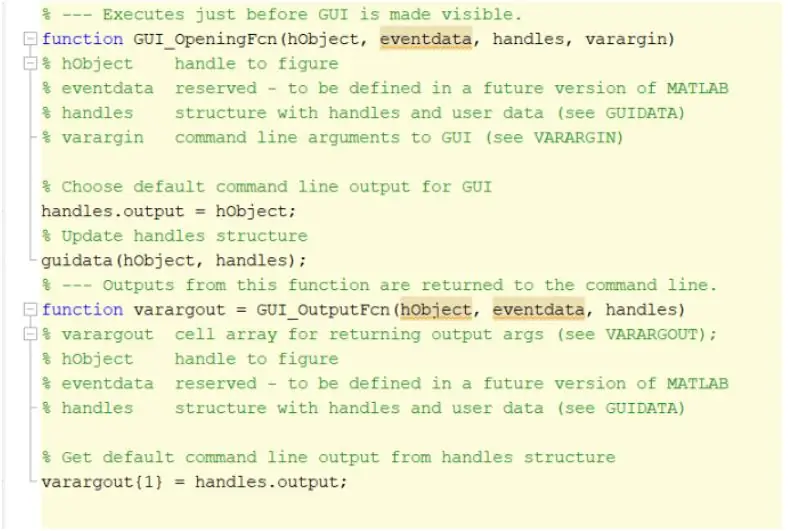
Die eerste stap sou wees om die grafiese gebruikerskoppelvlak, GUI, te skep en te begin. Dit kan gedoen word deur die gids in die opdragvenster in te tik, op enter te druk en 'n nuwe GUI te skep. Sodra hierdie stap voltooi is, kan u funksies begin skep soos byle, statiese teks, teks wysig en drukknoppies wat vertoon sal word sodra die program uitgevoer is en die gebruiker kan kommunikeer. Hierdie funksies kan deur die eiendomsinspekteur geredigeer en gemanipuleer word, maar die belangrikste kenmerk wat by die skep van hierdie funksies verander moet word, is die naam Tag. Dit is belangrik om die tagnaam van elke funksie wat geïmplementeer word, te verander, want dit sal ons toelaat om 'n herkenbare terugbelfunksie te skep. As u tevrede is met die uitleg van u GUI, kan u voortgaan met die laai van die DICOM -lêers wat in die GUI verskyn.
Stap 2: Laai en lees MRI -beelde in MATLAB
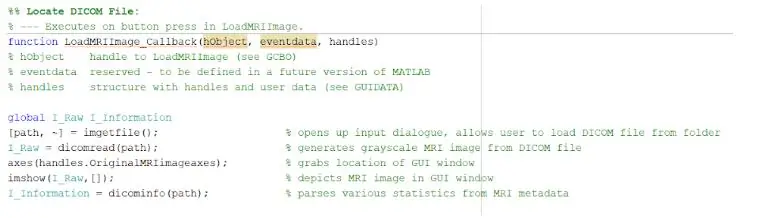
Om die DICOM -lêers te laai, moet u die terugbelfunksie wat met die knoppie 'Laai MRI -prent' gedruk word, initialiseer. Sodra dit voltooi is, moet u 'n globale veranderlike skep wat die beeld op die handvatsels sal vertoon waar u wil hê dat die oorspronklike MRI -prent vertoon moet word. Die MRI -skande -beelde wat van die databasis afgelaai word, is almal DICOM -geformateerde lêers wat in u MATLAB -gids gelaai moet word. Soek die lêer met behulp van imgetfile om dit in die program te laai. Die beelde word gelees met behulp van die ingeboude MATLAB -funksie 'dicomread', en die eerste rou beeld vir elke lêer word met imshow in die linker GUI -as ingebed.
Die ingeboude MATLAB -funksie 'dicominfo' is ook uiters handig om al die inligting van elke MRI -dicom -lêer aan te spreek. Ons gebruik hierdie funksie om al die beskrywende inligting van die pasiënte, soos hul geslag, ouderdom, gewig en lengte, uit te haal. Hierdie funksie bied u ook die stapelvolgorde wat nuttig is vir die implementering van die program binne die grafiese gebruikerskoppelvlak. Ons het veranderlikes geskep vir elk van die beskrywende inligting van die pasiënte wat vir die GUI gebruik sal word wanneer op die detect -knoppie gedruk word.
Stap 3: Beeldfiltering

Nadat die DICOM -lêer van die rou beeld gelaai en gelees is, moet die beeld van grysskaal omgeskakel word na 'n binarized vorm wat slegs uit swart en wit pixels bestaan. Ons gebruik die funksie 'imbinarize' om 'n binêre beeld uit die rou beeld te skep deur die beheer van aspekte van adaptiewe drempelwaarde teen sensitiwiteitswaarde van 0,59. Die standaard drempelgevoeligheidsfaktor, 0.5 was laag en kon nie die helderder kolle en kolle uit die beeld opspoor nie, dus het ons dit verhoog tot 0.59.
Die binarized beeld word dan verwerk deur middel van 'n mediaan filter met die funksie 'medfilt2' omdat die binarized beeld tweedimensioneel is. Ons stel elke uitvoerpixel in om die mediaanwaarde in die 5 x 5 -omgewing rondom die ooreenstemmende pixel in die ingevoerde beeld te bevat. Dit verminder die geraas en behou die rande in 'n 5 x 5 vierkant rondom elke pixel. Vervolgens pas ons 'n skuifvenster toe met 'strel' om 'n skyfvormige plat struktuurelement met 'n buurtradius van 2 te skep om elke sentrale, oorspronklike pixel in elke skyfbuurt te identifiseer. Ons het 'n skyfstruktuurelement gebruik omdat ons elke sirkelvlek en die pixels binne elke plek ontleed, dus is 'n skyfvormelement meer bruikbaar.
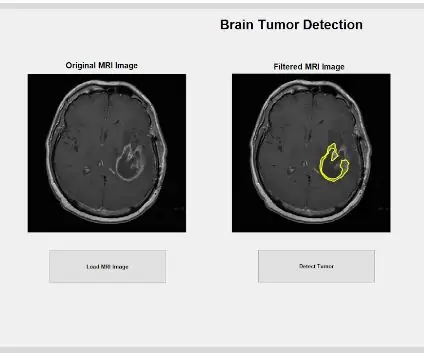
Sodra die prent gefiltreer is, kan dit skoongemaak word met die 'sluit' -funksie om die swart kolle tussen die gefiltreerde wit pixels in die prent te verwyder en al die gapings om dit te sluit. Die volledig verwerkte beeld kan dan in die tweede subplot van die vooraf toegewysde figuur geteken word, wat 'n vergelyking tussen die rou en gefiltreerde beeld moontlik maak.
Stap 4: Tumorisolasie deur elliptiese masker


Die ligpunte van die gewas kan dan van die hoof gefiltreerde beeld geïsoleer word deur 'n vooraf gegenereerde elliptiese masker. Om hierdie masker te skep, moet u die grootte van die oorspronklike, rou MRI-skanderingbeeld ken en die rye- en kolomlengte as x en y- koördinate gebruik, die middelpuntkoördinate vir die elliptiese toewys. Ons stel die y-as as 'n hoofas met 'n radius van 50 eenhede van die middel, en die klein as met 'n radius van 40 eenhede van die middel.
Ons het die MATLAB-funksie 'meshgrid' gebruik om 'n kartesiese vlak met tweedimensionele roosterkoördinate te genereer, gebaseer op die koördinate in vektore van 1 tot die lengte van die x-as, en van 1 tot die lengte van die y-as van die beeld. Col is 'n matriks waar elke ry 'n kopie van die x-as is, en Ry 'n matriks is waar elke kolom 'n afskrif van die y-as is. Die kartesiese rooster wat deur die koördinate Col en Row voorgestel word, het lengte (1: Y_Size) rye en lengte (1: X_Size) kolomme. Gebruik die indekse van Col en Row wat deur die kartesiese rooster gegenereer word om die vergelyking van ellips te bepaal, afhangende van die voorafbepaalde radius en middelkoördinate. Die elliptiese buitelyn kan nou gevul word met die wit pixels wat van gewasvlekke gevind word.
Deur gebruik te maak van die vooraf gegenereerde elliptiese masker, kan ons die spesifieke gewas wat u wil ontleed, uit die gefiltreerde beeld haal. Die elliptiese masker ontdek watter kolle logies binne die buitelyn van die ellips pas en aanvaar dit as 'n plek op die gefiltreerde beeld om as 'n gewas aanvaarbaar te wees. Die funksie 'bwareafilt' filtreer dan alle ander voorwerpe buite hierdie aangetaste tumor uit die beeld. Ons het 'n spesifieke venster van 500 by 4000 empiries gebruik, gebaseer op die afmetings van al die beelde. Daarna het ons 'n ander skuifvenster met 'strel' as 'n plat skyfvormige struktuurelement met 'n groter buurtradius van 6 aangebring om die gapings tussen elke sentrale wit pixel binne die opgespoorde tumor te sluit. Die opgespoorde gewasvlek word verder skoongemaak met 'imclose' om die swart pixels verder uit te skakel en al die gate met 'invulling' in te vul. Hierdie verwerkte gewas kan dan in die derde subplot in die vooraf toegewysde plot vertoon word om 'n vergelyking te bied tussen die geïsoleerde tumor en die oorspronklike en gefiltreerde beelde van die MRI -skandering.
Stap 5: Tumor -uiteensetting
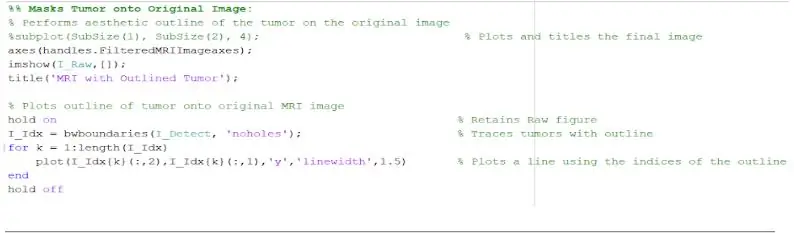
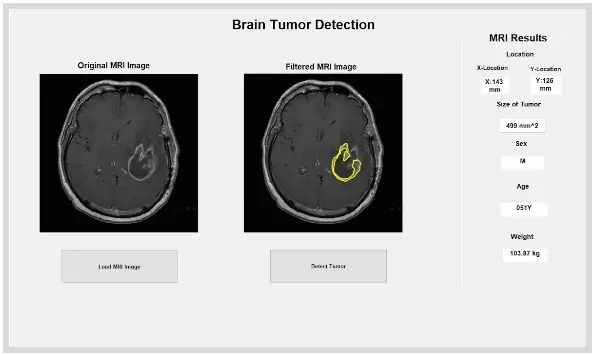
Noudat die gewas met die masker geïsoleer is, kan dit omlyn word en op die oorspronklike beeld vertoon word om die presiese ligging daarvan te toon. Om dit te kan doen, gebruik ons die funksie 'bwboundaries' om die gewas wat voorheen opgespoor is met 'n buitelyn op te spoor. Ons het die buitelyn gespesifiseer om nie die gate in die gewasvoorwerp te bevat soos dit uiteengesit word nie. Dit kan op die oorspronklike, rou beeld uitgebeeld word, met 'n 'vir' -lus wat die buitelyn rondom die gewas met die indekse van die lyn met 'n lynwydte van 1,5 pixels teken. Hierdie uiteensetting word dan op die rou beeld geteken, met die presiese grootte en ligging van die gewas, relatief tot die oorspronklike MRI -skandering.
Stap 6: Ontleding van die fisiese eienskappe van gewasse
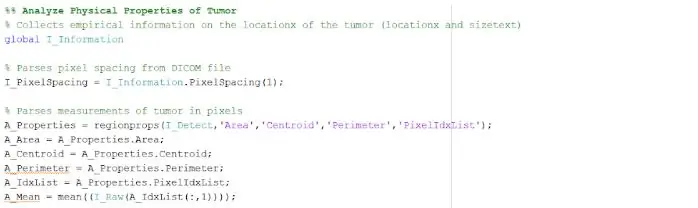
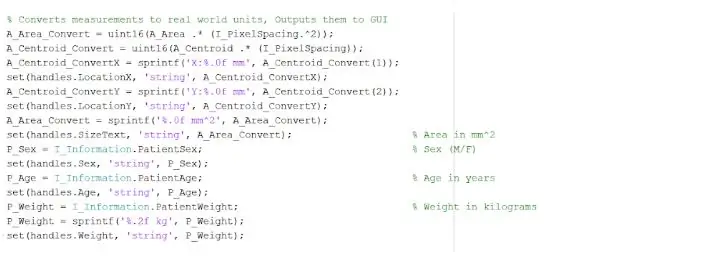
Die geïsoleerde en uiteengesit plek kan ons nuttige inligting gee oor die grootte, oppervlakte en ligging van die gewas. Ons het die 'regionprops' -funksie gebruik om die eienskappe van die gewas op te spoor wat betrekking het op oppervlakte, omtrek, sentroïede en die pixelindekswaarde. Hierdie pixelindekswaarde gee ons die regte wêreld -eenhede vir elke pixel van elke prent, uniek vir elke skandering. Hierdie eiendomme kan dan omgeskakel word in werklike eenhede van millimeters. Die empiriese inligting wat die program aan ons verskaf, is uniek vir elke MRI -skandering en is uiters handig om die grootte, ligging en tipe tumor te bepaal wat die gebruikers kan analiseer en in die grafiese gebruikerskoppelvlak kan inkorporeer.
Aanbeveel:
Arduino Nano -klok met aanpasbare helderheid met behulp van prototipe PCB van NextPCB.com: 11 stappe

Arduino Nano -klok met aanpasbare helderheid met behulp van prototipe PCB van NextPCB.com: Almal wou 'n horlosie hê wat tyd en datum saam wys. In hierdie projek sal ek u wys hoe u 'n arduino -nano -klok met aanpasbare helderheid kan bou met behulp van RTC en 'n ontwerp PCB van NextPCB
Beheer Raspberry Pi met behulp van Matlab: 5 stappe

Beheer Raspberry Pi met behulp van Matlab: Hey, hierdie tutoriaal handel oor die beheer van jou koplose framboos pi met behulp van matlab. Miskien moet u matlab in die nuutste weergawe installeer vir ondersteuning vir 'n nuwer framboos -pi -bord
Beheer oor die hele wêreld met behulp van internet met behulp van Arduino: 4 stappe

Beheer oor die hele wêreld met behulp van internet met behulp van Arduino: Hallo, ek is Rithik. Ons gaan 'n internetbeheerde LED maak met u telefoon. Ons gaan sagteware soos Arduino IDE en Blynk gebruik. Dit is eenvoudig, en as u daarin geslaag het, kan u soveel elektroniese komponente beheer as wat u wil
Monitor versnelling met behulp van Raspberry Pi en AIS328DQTR met behulp van Python: 6 stappe
Monitering van versnelling met behulp van Raspberry Pi en AIS328DQTR Gebruik van Python: Versnelling is eindig, volgens ek volgens sommige wette van die fisika.- Terry Riley 'n Jagluiperd gebruik ongelooflike versnelling en vinnige spoedveranderinge wanneer hy jaag. Die vinnigste wesens aan wal gebruik af en toe sy hoogste tempo om prooi te vang. Die
Hoe om van 0 tot 99 te tel met behulp van 8051 mikrobeheerder met 7 segmentweergawe: 5 stappe (met foto's)
Hoe om van 0 tot 99 te tel Met behulp van 8051 mikrobeheerder met 7 -segmentweergawe: Hallo almal, in hierdie tutoriaal gaan ons u vertel hoe om van 0 tot 99 te tel met twee 7 -segment -skerms
