
INHOUDSOPGAWE:
- Outeur John Day day@howwhatproduce.com.
- Public 2024-01-30 07:25.
- Laas verander 2025-01-23 12:53.

ThinkBioT is ontwerp om 'Plug and Play' te wees, met Edge TPU -versoenbare TensorFlow Lite -modelle.
In hierdie dokumentasie gaan ons oor die maak van spektrogramme, die opmaak van u data en die gebruik van Google AutoML.
Die kode in hierdie tutoriaal sal in bash geskryf word, en sal dus multi-platform versoenbaar wees.
Afhanklikes
- Maar voordat u begin, moet u Sox 'n opdragreël -klankprogram installeer wat versoenbaar is met Windows-, Mac- en Linux -toestelle.
- As u op 'n Windows -toestel is, is die maklikste manier om bash -scripts uit te voer via Git, so ek sal dit aanbeveel en aflaai en installeer, aangesien dit op baie maniere nuttig is,
- Gebruik u gunsteling redakteur vir die redigering van kode, of installeer NotePad ++ vir Windows of Atom vir ander bedryfstelsels.
** Raadpleeg die Google Coral -dokumentasie as u 'n bestaande TensorFlow -model het of u wil leer met 'n bestaande model.
Stap 1: Stel 'n Google Cloud Storage Bucket op
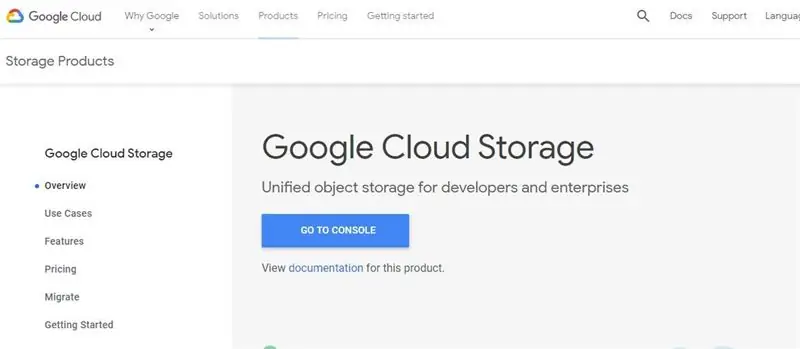
1. Meld aan by u gmail -rekening (of skep een as u nie 'n Google -rekening het nie)
2. Gaan na die projekkeuse -bladsy en maak 'n nuwe projek vir u model- en spektrogramlêers. U moet fakturering in staat stel om verder te vorder.
3. Besoek https://cloud.google.com/storage/ en druk die knoppie skep bo -aan die bladsy.
4. Voer die gewenste emmernaam in en skep die emmer met standaardinstellings.
Stap 2: Formateer u data en skep databasis Csv
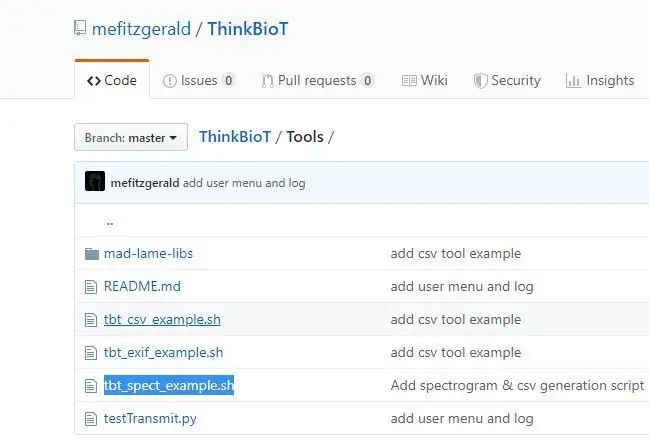
Ek het 'n nuttige script ontwerp om u dataset.csv -lêer te skep wat nodig is om u model te skep. Die dataset -lêer koppel die beelde in u emmer aan die etikette in die dataset.
1. Laai die ThinkBioT -bewaarplek af van GitHub en
2. Kopieer die tbt_spect_example.sh lêer van die gids Tools na 'n nuwe gids op u lessenaar.
3. Voeg die klanklêers by wat u in u model wil gebruik, en plaas dit in gidse met hul etiket (dws waarin u dit wil rangskik. As u byvoorbeeld honde of katte wil identifiseer, kan u 'n vouer hê hond, met blafgeluide OF map met die naam kat met katgeluide ens.
4. Maak die tbt_spect_example.sh oop met Notepad ++ en vervang "jou boknaam" in reël 54 met die naam van jou Google Storage Bucket. As u emmer byvoorbeeld myModelBucket genoem word, word die reël verander na
bucket = "gs: // myModelBucket/spectro-data/"
5. Begin die kode deur die volgende in u Bash-terminale in te tik, die kode sal loop en u csv-lêer met etikette en 'n gids met die spektro-data op u tafelblad met die resultate spektrogramme skep.
sh tbt_spect_example.sh
Stap 3: Laai u spektrogramme op na u emmer
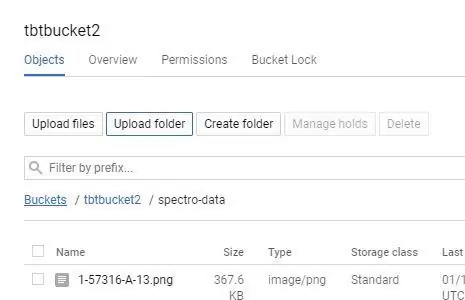
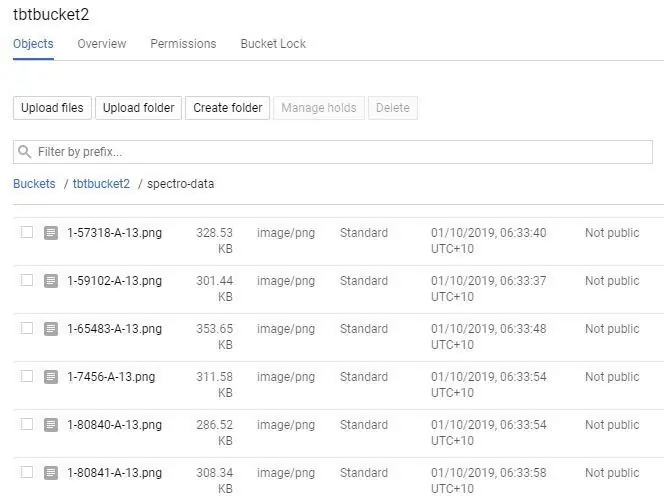
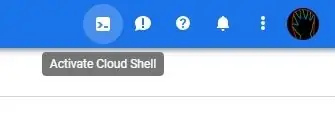
Daar is 'n paar maniere om na Google Storage op te laai; die maklikste is om die lêer direk te laai;
1. Klik op u emmernaam op u Google Storage -bladsy.
2. Kies die "UPLOAD FOLDER" knoppie en kies u "spectro-data/" gids wat in die laaste stap geskep is.
OF
2. As u 'n groot hoeveelheid lêers het, kan u die "spectro-data/" -gids handmatig skep deur "CREATE FOLDER" te selekteer, navigeer dan in die gids en kies "UPLOAD FILES". Dit kan 'n uitstekende opsie wees vir groot datastelle, aangesien u die spektrogramme in afdelings kan oplaai, selfs met verskeie rekenaars om die oplaaisnelheid te verhoog.
OF
2. As u 'n gevorderde gebruiker is, kan u ook oplaai via Google Cloud Shell;
gsutil cp spectro-data/* gs: // your-bucket-name/spectro-data/
U behoort nou 'n emmer vol mooi spektrogramme te hê!
Stap 4: Laai u databasis Csv
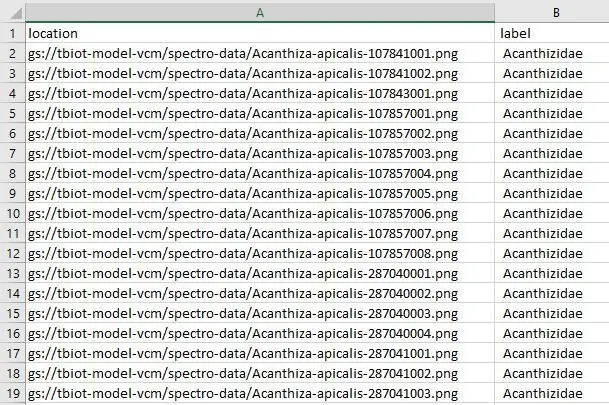
Nou moet ons die model-labels.csv-lêer oplaai na u 'spectro-data/' gids in Google Storage, dit is in wese dieselfde as die laaste stap; u laai net 'n enkele lêer op in plaas van baie.
1. Klik op u emmernaam op u Google Storage -bladsy.
2. Kies die "UPLOAD FILE" -knoppie en kies u model-labels.csv-lêer wat u vroeër geskep het.
Stap 5: Skep 'n datastel
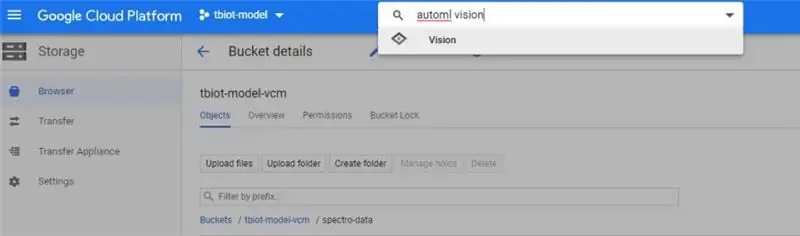
1. Eerstens moet u die AutoML VIsion API vind, dit kan 'n bietjie lastig wees! Die maklikste manier is om 'outomatiese visie' te soek in die soekbalk van u Google Cloud -berging (op die foto).
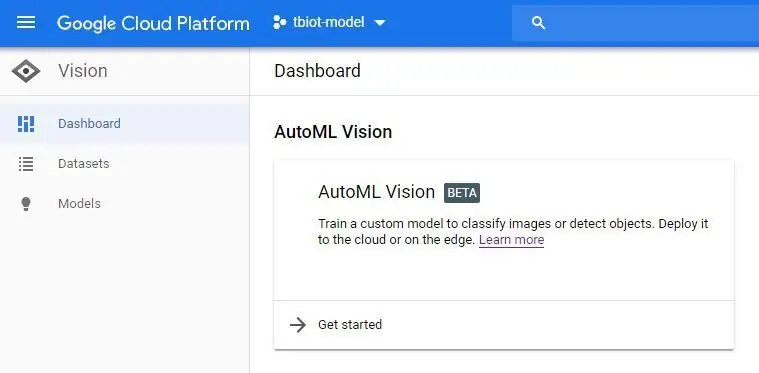
2. Sodra u op die API -skakel geklik het, moet u die API aktiveer.
3. Nou sal u in die AutoML Vision Dashboard (op die foto) klik op die nuwe datastelknoppie en kies Enkel etiket en die 'Kies 'n CSV -lêer' opsie. U sal dan die skakel na u model-labels.csv-lêer in u stooremmer insluit. As u hierdie tutoriaal gevolg het, sal dit soos hieronder beskryf word
gs: //yourBucketName/spectro-data/model-labelsBal.csv
4. Druk dan op voortgaan om u datastel te skep. Dit kan 'n rukkie neem om dit te skep.
Stap 6: Skep u AutoML -model
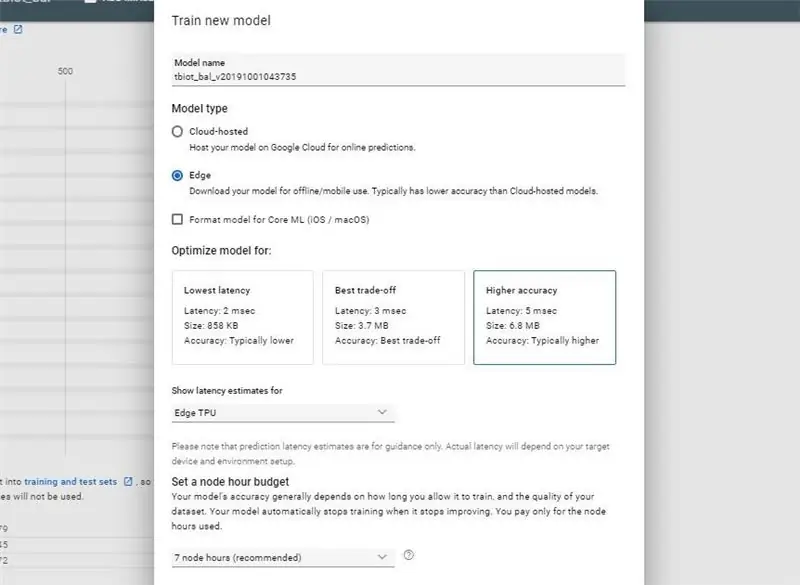

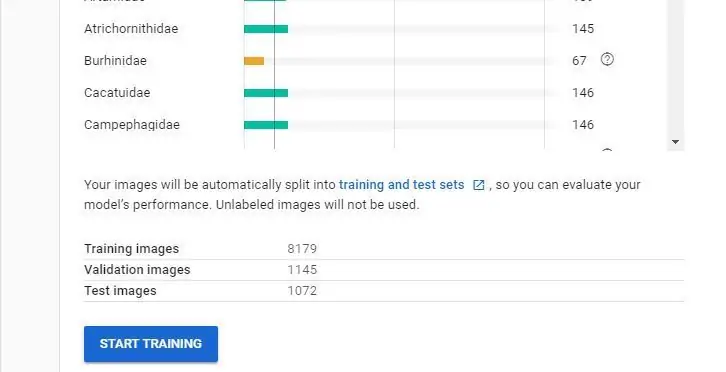
Sodra u u e -pos ontvang het waarin u weet dat u datastel geskep is, is u gereed om u nuwe model te skep.
- Druk die TRAIN -knoppie
- Kies modeltipe: Rand- en modelvertragingsberamings: Edge TPU en laat die ander opsies aanvanklik as standaard, moeilik, waarmee u later sal kan eksperimenteer.
- Nou sal u model leer, dit sal 'n rukkie neem en u sal 'n e -pos ontvang wanneer dit gereed is om af te laai.
Let wel: as u 'n oefenknoppie nie beskikbaar is nie, het u moontlik probleme met u datastel. As u minder as 10 van elke klas (etiket) het, sal die stelsel u nie toelaat om 'n model op te lei nie, sodat u moontlik ekstra beelde moet byvoeg. Dit is die moeite werd om na die Google AutoML -video te kyk as u verduideliking benodig.
Stap 7: Toets u model
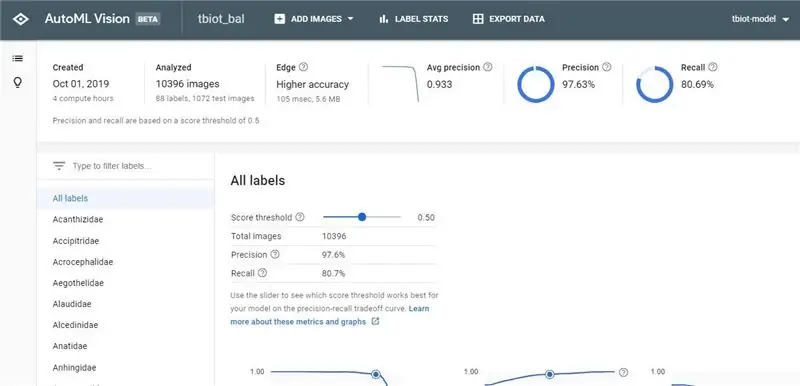
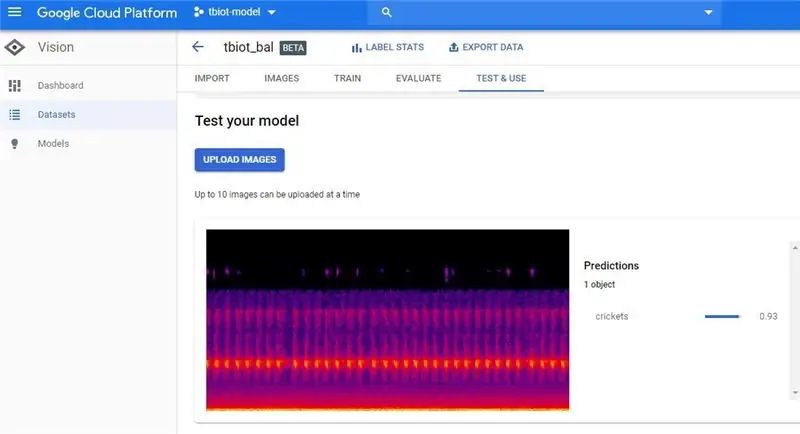
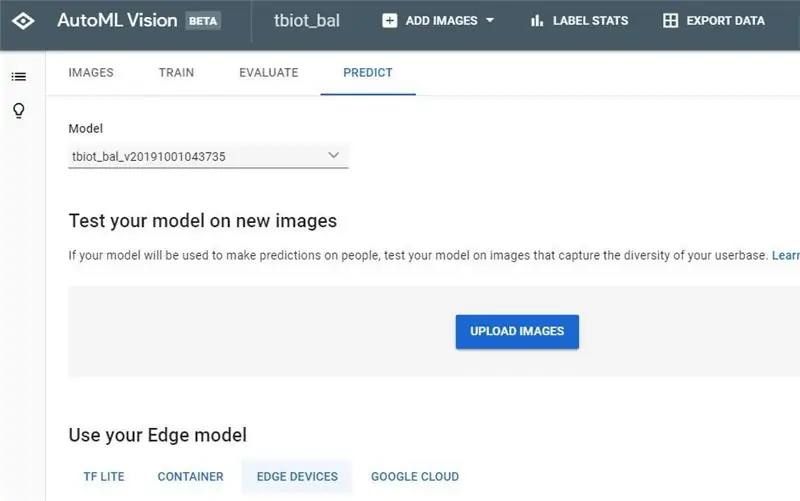
Sodra u u model -voltooiings -e -pos ontvang het, klik op die skakel om terug te keer na die AutoML Vision API.
1. Nou sal u u resultate en die verwarringsmatriks vir u model kan sien.
2. Die volgende stap is om u model te toets, na 'TEST & GEBRUIK' of 'VOORSPELLING' te gaan, vreemd dat daar twee gebruikers -GUI's is, wat ek albei voorgestel het, maar beide opsies het dieselfde funksie.
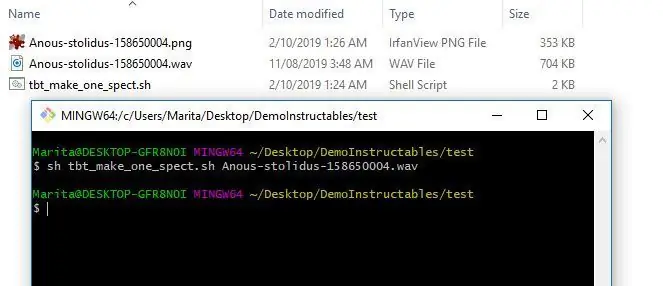
3. Nou kan u 'n toets spektrogram oplaai. Om 'n enkele spektrogram te maak, kan u die tbt_make_one_spect.sh -program van die ThinkBioT Github gebruik. Laai dit eenvoudig in 'n gids met die wav wat u in 'n spektrogram wil omskep, maak 'n Git Bash -venster (of terminale) oop en gebruik die onderstaande kode en vervang u lêernaam.
sh tbt_make_one_spect.sh yourWavName.wav
4. Laai nou eenvoudig die spektrogram op en kyk na u resultaat!
Stap 8: Installeer u model in ThinkBioT
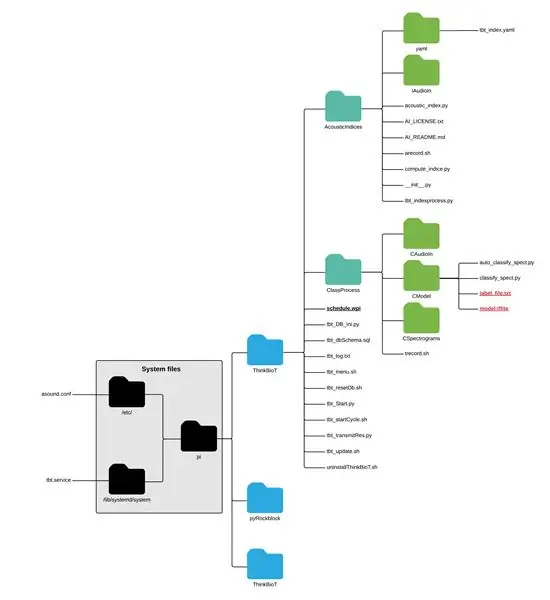
Om u nuwe blink model te gebruik, moet u die model en die txt -lêer in die CModel -lêergids laat val;
pi> ThinkBioT> ClassProcess> CModel
Nou is u gereed om ThinkBioT te gebruik:)
** NB ** As u u model buite die ThinkBioT-raamwerk gebruik, moet u u etiketdokument wysig as byvoegsels aan die begin van elke reël, aangesien die nuutste tflite-tolke ingeboude "leesetikette" -funksie veronderstel dat hulle daar is. Ek het 'n pasgemaakte funksie in die ThinkBioT -raamwerk, classify_spect.py, geskryf as 'n werk wat u in u eie kode kan gebruik:)
def ReadLabelFile (file_path):
teller = 0 met oop (file_path, 'r', encoding = 'utf-8') as f: lines = f.readlines () ret = {} vir reël in reëls: ret [int (counter)] = line.strip () teller = teller + 1 retourret
Aanbeveel:
Bewegings geaktiveerde cosplay -vleuels met behulp van Circuit Playground Express - Deel 1: 7 stappe (met foto's)

Motion Activated Cosplay Wings Using Circuit Playground Express - Deel 1: Dit is deel een van 'n tweedelige projek, waarin ek jou my proses vir die maak van 'n paar outomatiese sprookjesvlerke gaan wys. Die eerste deel van die projek is die meganika van die vlerke, en die tweede deel maak dit draagbaar en voeg die vlerke by
Outomatiseer 'n kweekhuis met LoRa! (Deel 2) -- Gemotoriseerde vensteropening: 6 stappe (met foto's)

Outomatiseer 'n kweekhuis met LoRa! (Deel 2) || Gemotoriseerde vensteropening: In hierdie projek sal ek u wys hoe ek 'n gemotoriseerde vensteropening vir my kas gemaak het. Dit beteken dat ek u sal wys watter motor ek gebruik het, hoe ek die werklike meganiese stelsel ontwerp het, hoe ek die motor bestuur en laastens hoe ek 'n Arduino LoRa gebruik het
Deel 1. ThinkBioT outonome bio-akoestiese sensor hardeware Bou: 13 stappe

Deel 1. ThinkBioT outonome bio-akoestiese sensor hardeware bou: ThinkBioT het ten doel om 'n sagteware- en hardeware raamwerk te bied, ontwerp as 'n tegnologiese ruggraat om verdere navorsing te ondersteun, deur die besonderhede van data-insameling, voorafverwerking, data-oordrag en visualiseringstake te hanteer navorser
Hoe om u eie windmeter te bou met behulp van rietskakelaars, saaleffek sensor en 'n paar stukkies op Nodemcu - Deel 2 - Sagteware: 5 stappe (met foto's)

Hoe om u eie windmeter te bou met behulp van rietskakelaars, saal -effektsensor en 'n paar stukkies op Nodemcu - Deel 2 - sagteware: inleiding Dit is die opvolger van die eerste pos "Hoe om u eie windmeter te bou met behulp van rietskakelaars, saal -effek sensor en 'n paar stukkies" op Nodemcu - Deel 1 - Hardeware " - waar ek wys hoe om die windsnelheid en rigtingmeting saam te stel
Hoe om u eie windmeter te bou met behulp van rietskakelaars, saal -effek sensor en 'n paar stukkies op Nodemcu. - Deel 1 - Hardeware: 8 stappe (met foto's)

Hoe om u eie windmeter te bou met behulp van rietskakelaars, saal -effek sensor en 'n paar stukkies op Nodemcu. - Deel 1 - Hardeware: Inleiding Sedert ek begin het met die studie van Arduino en die Maker Culture, het ek graag nuttige toestelle gebou met rommel- en afvalstukkies, soos botteldoppe, stukke PVC, blikkies, ens. Ek gee graag 'n tweede lewe vir enige stuk of 'n maat
